Home / Domaines défis / RCanGéCO / VirusSeq
VirusSeq

L’initiative VirusSeq du RCanGéCO procède au séquençage de quelque 150 000 échantillons viraux de personnes ayant reçu un diagnostic positif à la COVID-19 et veille à une coordination accrue du séquençage du virus et de la collecte des données cliniques/épidémiologiques connexes entre les provinces et à l’échelle internationale.
Cet effort permet au Canada de suivre comment le virus SRAS-CoV-2, à l’origine de la COVID-19, change et se propage, obtenant ainsi des données clés sur les tendances de la transmission du virus et ses changements qui peuvent nuire à son dépistage ou à l’efficacité des traitements ou des vaccins.
Comme le SRAS-CoV-2 se propage d’une personne à une autre, il continue de muter. Il est possible de suivre ces mutations au moyen du séquençage du génome du virus. Ces mutations peuvent ensuite être utilisées pour produire un « arbre généalogique » de ce virus et de ses « descendants », ce qui aide les chercheurs à établir des liens entre les cas apparentés. Les modifications du virus peuvent aussi influencer la gravité de la maladie ou sa capacité à se propager. Les données des séquences des génomes viraux sont donc devenues un outil essentiel pour les représentants de la santé publique qui peuvent ainsi mieux suivre à la fois les changements et la propagation du virus.
Chronologie du virus
Novembre 2019
Le coronavirus humain SARS-CoV-2, le virus à l’origine de l’éclosion de COVID-19, a fait son apparition à la fin de novembre 2019 et en quatre mois, il était devenu pandémique, se propageant à 177 pays au moins et entraînant l’une des interruptions les plus importantes des activités humaines de l’histoire moderne.
Janvier 2020
La publication rapide, le 10 janvier 2020, de la première séquence génomique s’est avérée indispensable aux efforts mondiaux de lutte contre la COVID-19 et ses répercussions. Ce séquençage a permis non seulement d’amorcer le développement de vaccins et de médicaments antiviraux, mais il a aussi permis de concevoir des tests diagnostiques moléculaires importants et de les partager dans le mois où la maladie a été déclarée à l’OMS. Ces tests diagnostiques ne peuvent toutefois que confirmer si une personne a la COVID-19 ou pas. Les séquences génomiques déterminent de plus comment les cas sont liés entre eux, de sorte qu’on peut les utiliser pour retrouver l’origine du virus et déterminer également comment il se propage à l’échelle nationale et internationale.
De février 2020 à aujourd’hui
Les séquences génomiques, échangées rapidement avec les données contextuelles, ont permis de confirmer les introductions du virus au-delà des frontières et de reconnaître les cas de transmission communautaire, ce qui a orienté les politiques de lutte contre les éclosions. Les efforts soutenus de séquençage génomique sont indispensables à la modélisation de l’irruption et de la propagation du virus à l’échelle locale. À mesure que le nombre de cas de COVID-19 attribuables à la transmission communautaire augmentera, les enquêtes épidémiologiques et le suivi dépendront de plus en plus des données génomiques qui serviront de données probantes pour le dépistage des grappes de cas. Il est également essentiel d’assurer une surveillance continue basée sur le génome après la réduction du nombre de cas pour définir et contrôler les sources de grappes qui font craindre une deuxième vague épidémique.
L’avenir
Les changements dans les séquences génomiques, associés à la gravité de la maladie ou un jour à la résistance aux médicaments, peuvent aussi être suivis et orienter éventuellement la gestion des patients à mesure que le virus évoluera.
Cliquer ici pour en savoir plus sur la gouvernance du projet VirusSeq du RCanGéCO.
pourquoi séquencer ce virus?
Déterminer et suivre les tendances de la transmission à l’échelle régionale, provinciale, nationale et internationale
Faciliter le dépistage de nouvelles grappes de cas/éclosions
Découvrir les caractéristiques évolutives du virus qui peuvent influencer
• Les méthodes de dépistage (CRP, sérologie)
• Les résultats cliniques (gravité de la souche) et la transmission
• L’efficacité des mesures en soins de santé, les traitements et les vaccins

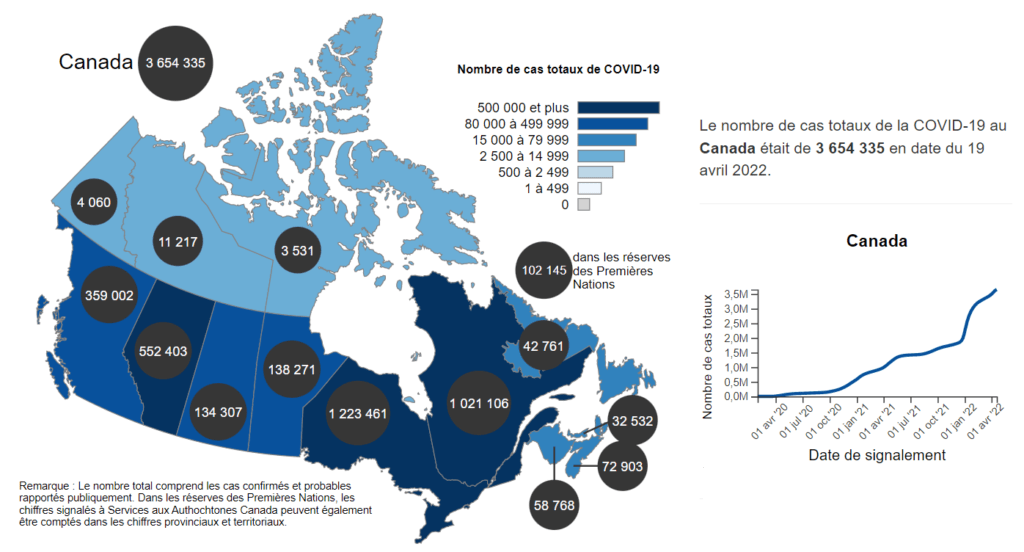
Coronavirus disease (COVID-19): Outbreak update
See the current situation with total cases, active cases, recovered cases, tests performed or deaths in Canada over time.
Ressources
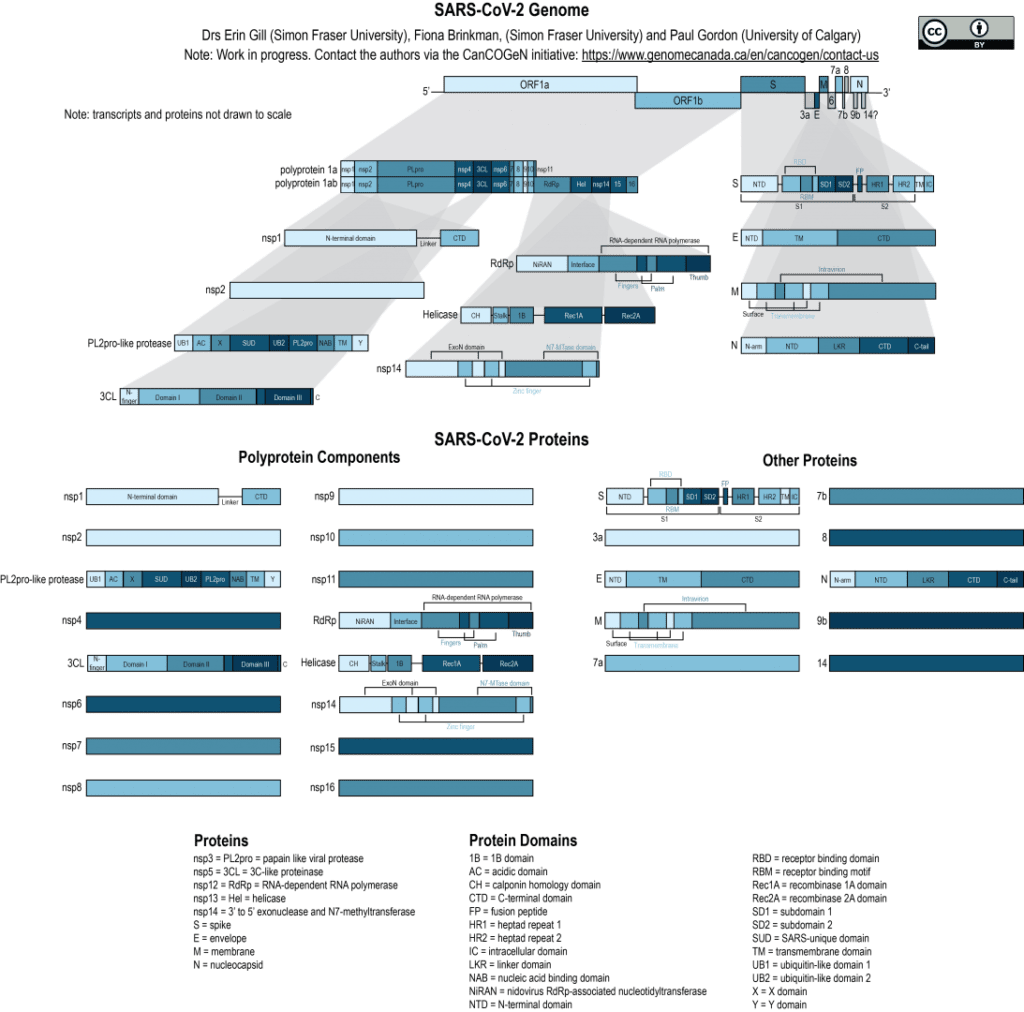
Schéma du génome du SRAS-CoV-2 qui illustre la complexité des protéines qui y sont codées. Le génome contient deux cadres de lecture ouverts (ORF1a et ORF1b) qui code les polyprotéines 1a et 1b. Ces dernières sont clivées en leurs éléments protéiques individuels après la traduction. Le génome code aussi d’autres protéines, dont les protéines structurales (en particulier la protéine Spike/protéine S/gène), qui sont traduites séparément. Chaque protéine codée par le génome du SRAS-CoV-2 est indiquée individuellement et les différents domaines fonctionnels/structurels sont annotés dans les protéines choisies.
Ressources
Schéma du génome du SRAS-CoV-2 qui illustre la complexité des protéines qui y sont codées. Le génome contient deux cadres de lecture ouverts (ORF1a et ORF1b) qui code les polyprotéines 1a et 1b. Ces dernières sont clivées en leurs éléments protéiques individuels après la traduction. Le génome code aussi d’autres protéines, dont les protéines structurales (en particulier la protéine Spike/protéine S/gène), qui sont traduites séparément. Chaque protéine codée par le génome du SRAS-CoV-2 est indiquée individuellement et les différents domaines fonctionnels/structurels sont annotés